Bonjour tout le monde !
Je viens vers vous en désespoire de cause. Je fais des tests antibiotiques sur des bacteries et je ne sais pas si c'est mes bactéries qui ont une dent contre moi ou si je suis a coté de la plaque mais impossible d'appliquer le model mathématique de mon protocole à mes courbes.
J'explique rapidement mon protocole :
Sur microplaque je mesures l'absorbance (tubidité) de solutions bacteriennes en présence de différentes concentrations d'une substance à propriété antibiotique
Il a donc sur chaque plaque (6 réplicats) un témoin négatif (ma bacterie toute seule dans son LB), un témoin positif (gentamycine haute concentration) et ma substance en differentes concentration (avec pour chaque concentration un blanc sans bacteries). Je mesure sur spectro toute les 1/2h environs et fait une jolie courbe do en fonction du temps.
courbe5.png
A priori là comme ça (pour celles là du moins) je les trouve belles les courbes.
Ensuite je suis sencée faire une regression eponentielle sur les courbes de type y=ae^(µx) ou µ est mon taux de croissance puis je calcule le taux d'inhibition : 100-((µsubstance/µtemoin -)*100) et fais une courbe dose réponse %inhibition en fonction de la concentration.
Le problème c'est que mes taux de croissance calculés sont totalement bidon. Alors que visuellement y'a pas photo je me retrouve avec des µ substances plus grand que celui du témoin donnée5.png
J'ai essayé toute sorte de modeles mathématiques mais à part changer mon protocole je ne sais pas comment comparer mes courbes entre elles autre que visuellement.
helps please
-----



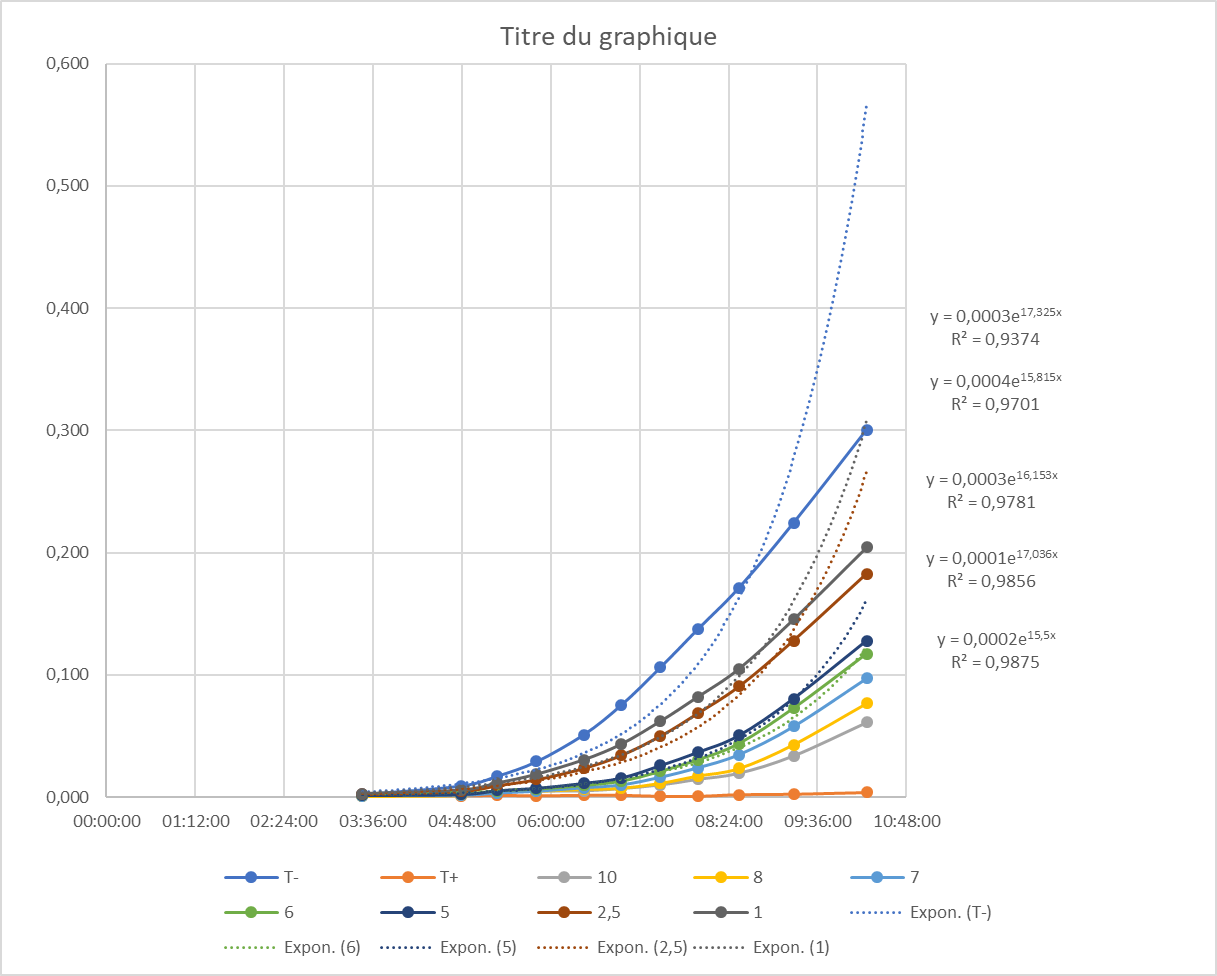 on voit d'ailleur assez bien que les courbes de regression ont l'air à coté de la plaque. Est-ce que je n'ai pas mesuré assez longtemps ? Est-ce qu'il y a un autre moyen à partir de ces courbes de calculer le % d'inhibition ?
on voit d'ailleur assez bien que les courbes de regression ont l'air à coté de la plaque. Est-ce que je n'ai pas mesuré assez longtemps ? Est-ce qu'il y a un autre moyen à partir de ces courbes de calculer le % d'inhibition ?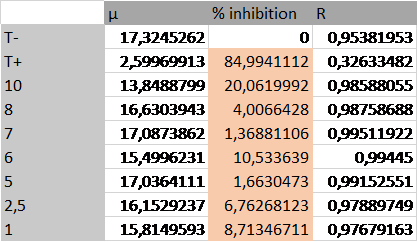

 .
.
