Bonjour à tous,
Je suis actuellement en L2-science de la vie et dans le cadre de mon UE génétique moléculaire je dois présenter un exposé sur le gène de mon choix. J'ai choisi de présenter le gène gh-25 qui est un gène qui est passionnant de par sa capacité à passer de taxons en taxons. Ce gène code pour une protéine: la gh-25 muramidase qui permet d'habitude aux bactéries de se reproduire en se scindant en deux et leur permet aussi de remodeler leur structure. Sauf que certains virus, certaines plantes, des champignons et des insectes ont "volé" ce gène pour l'utiliser contre les bactéries. Un sorte de gène antibiotique.
Pour pouvoir mener à bien mon exposé je me suis interessé à cet article: http://lens.elifesciences.org/04266/index.html#figures.
Ceci dit, l'anglais n'aidant pas il y a quelque chose que je n'arrive pas à comprendre ni à concevoir, c'est ce schéma:
je vous joins la légende :Et voilà là je ne comprend plus, que représente les barres composées des flèches ? Ce sont des chromosomes ? des gènes ? mais si ce sont des gènes ça voudrais dire que le gh-25 muramidase se serait pas un gène ?Architecture of HGT candidates and surrounding genes.
Each arrow represents an open reading frame transcribed from either the plus strand (arrow pointing right) or the minus strand (arrow pointing left). The color of the arrow indicates the taxa the gene is found in based on its closest homologs. Black = Eubacteria, purple = virus, red = Archaea, green = Plantae, Orange = Fungi, Blue = Insecta, white = no known homologs, dashed line = present in multiple domains. The length of the arrows and intergenic regions are drawn to scale except where indicated with broken lines. The four paralogs of the lysozyme in S. moellendorffii occur on two genomic scaffolds with light green bands connecting homologous genes. Vertical arrows indicate the location of introns in the A. oryzae and A. pisum lysozymes. Abbreviations: Lys: lysozyme, gpW = phage baseplate assembly protein W, SH3: Src homology domain 3, App-1 = ADP-ribose-1”-monophosphatase, PRT = phosphoribosyltransferase, LD = leucoanthocyanidin dioxygenase; IMP = integral membrane protein. A protein diagram for each lysozyme is drawn to scale with the light gray regions highlighting a conserved protein domain. *A. pisum diagram is based on Acyr_1.0 assembly and transcription data (Nikoh et al., 2010); the annotation in Acyr_2.0 is different.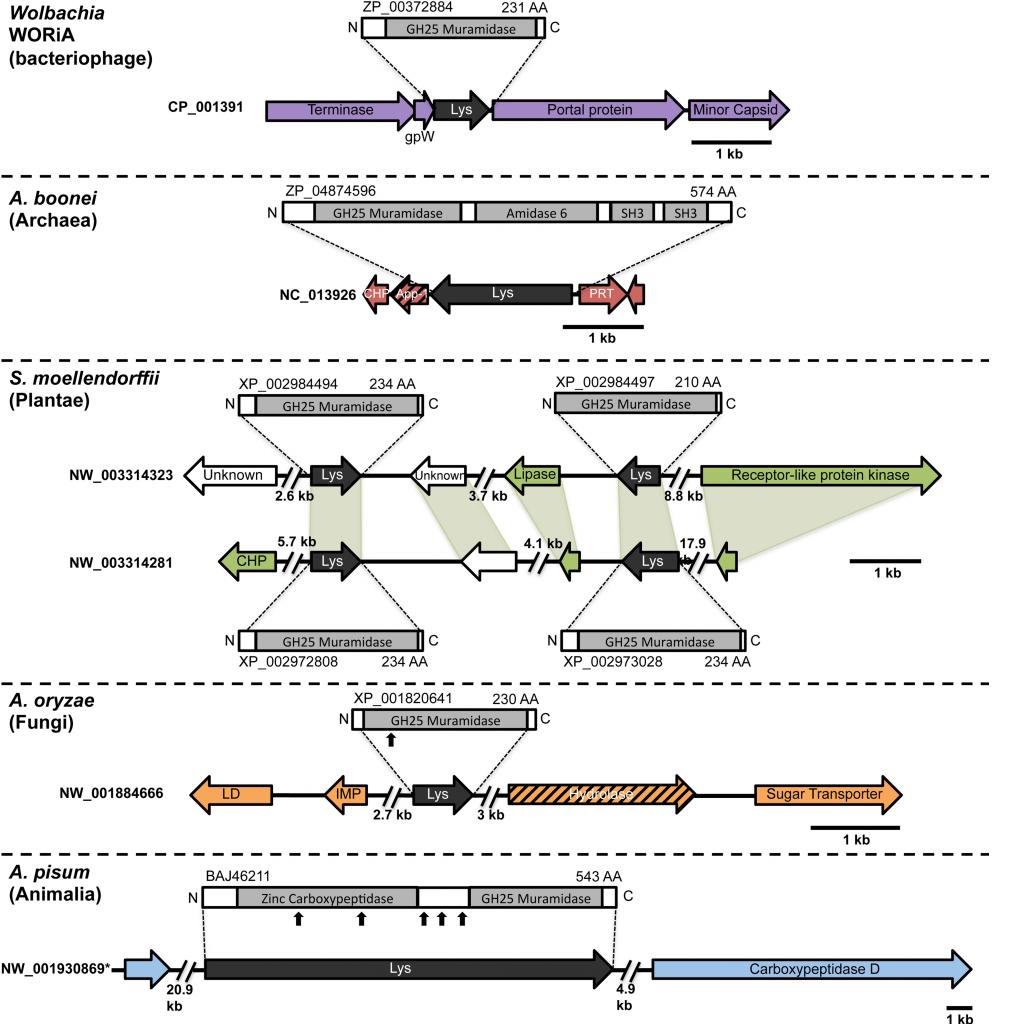
Je ne comprend pas nn plus le terme "plus strand" et "minus strand", à quel moment parle t'on de brin positif et de brin négatif ? C'est le brin initial et le brin complémentaire ?.
De plus il est écris que le couleur de la flèche indique le taxon dans lequel le gène a été trouvé en se basant sur son homologue le plus proche et là je ne pige pas non plus. Chaque gène représenté ne correspond pas à une espèce et un individu particulier ?
J'ai vraiment compris le principe du gène et comment en gros on a pu prouver le lien de parenté entre les différents gènes et les gènes des bactéries présentent dans les même niches écologiques mais j'ai vraiment du mal avec ce schéma, même les numéros écris sur la gauche je ne comprend pas à quoi ils font référence. Si quelqu'un doué en génétique, en anglais ou les deux pourrait éclairer ma lanterne parce que je suis vraiment perdu.
Je vous remercie et vous souhaite à tous une bonne soirée,
Xavier
-----




