Bonjour,
j'ai un exercice en Biologie moléculaire que je ne comprends vraiment pas et qui est crucial pour ce chapitre. Le voici:
QUESTIONS:
1. L’ADN génomique d’une bactérie marine récemment isolée de sédiments a été extrait et purifié. Ce génome a ensuite été soumis à un séquençage suivant le méthode de Sanger.
a) Résumer en vous appuyant sur un schéma le principe de la méthode de séquençage utilisée.
Ce séquençage a mis en évidence la présence, au sein du génome bactérien, d’un gène C codant visiblement pour une enzyme de dégradation de la chitine (polysaccharide de constitution des carapaces des crustacés, des coquilles de céphalopodes,…).
Une équipe de chercheur souhaite produire de manière recombinante cette enzyme dénommée . Pour cela et en utilisant un couple d’amorces adaptées, une première étape d’amplification par PCR du gène C est entreprise. La séquence nucléotidique partielle du gène C se trouve en annexe 1.
b) Quelles amorces utiliseriez-vous pour amplifier le gène C en sachant que le clonage dans le vecteur d’expression sera réalisé en utilisant l’enzyme de restriction EcoRI
(taille des amorces attendues : 20 nucléotides; séquence EcoRI : GAATTC) ?
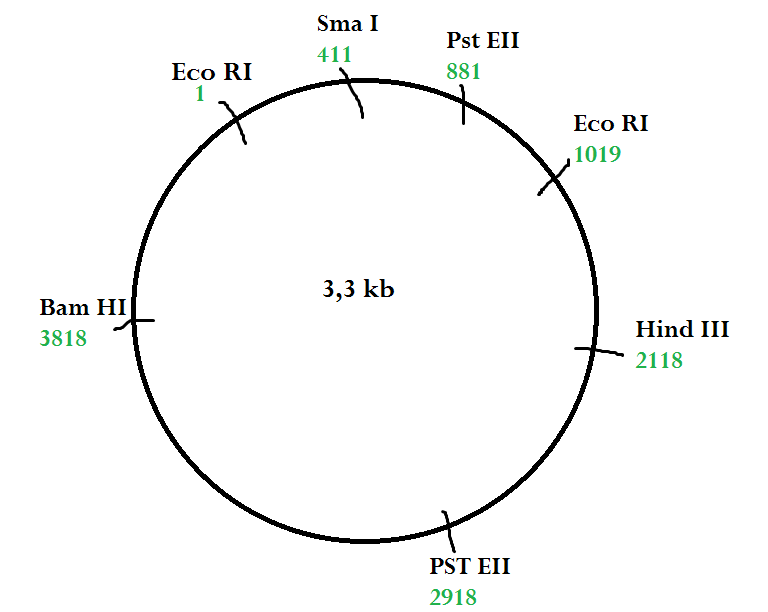
2. Le produit de PCR obtenu a été digéré et ligué dans un vecteur d’expression de 3,3 kb (pPICZA). Pour vérifier le clonage, différentes digestions ont été réalisées :
a) Les vecteurs pPICZA recombinant et non recombinant ont été digérés par les enzymes de restriction EcoRI ou PstEII. Compléter le gel d’agarose présenté en annexe 2 en dessinant pour chaque piste (1 à 4) les bandes correspondantes aux profils de restriction attendus.
b) Question bonus : Le vecteur recombinant a ensuite été digéré par les deux enzymes simultanément. Compléter le gel d’agarose (piste 5).
Mes réponses :
Pour la 1)a), pas de problème.
Pour la 1)b), j'utiliserai les amorces 5' ATGAGCATTTGTTATCACGA 3' et 3' AATAAGAAGGAAACGAAAGT 5'
C'est pour la 2)a) que ça se complique... Si quelqu'un peut m'expliquer
Merci d'avance !
-----