Bonjour,
j'apprends à utiliser BLASTx.
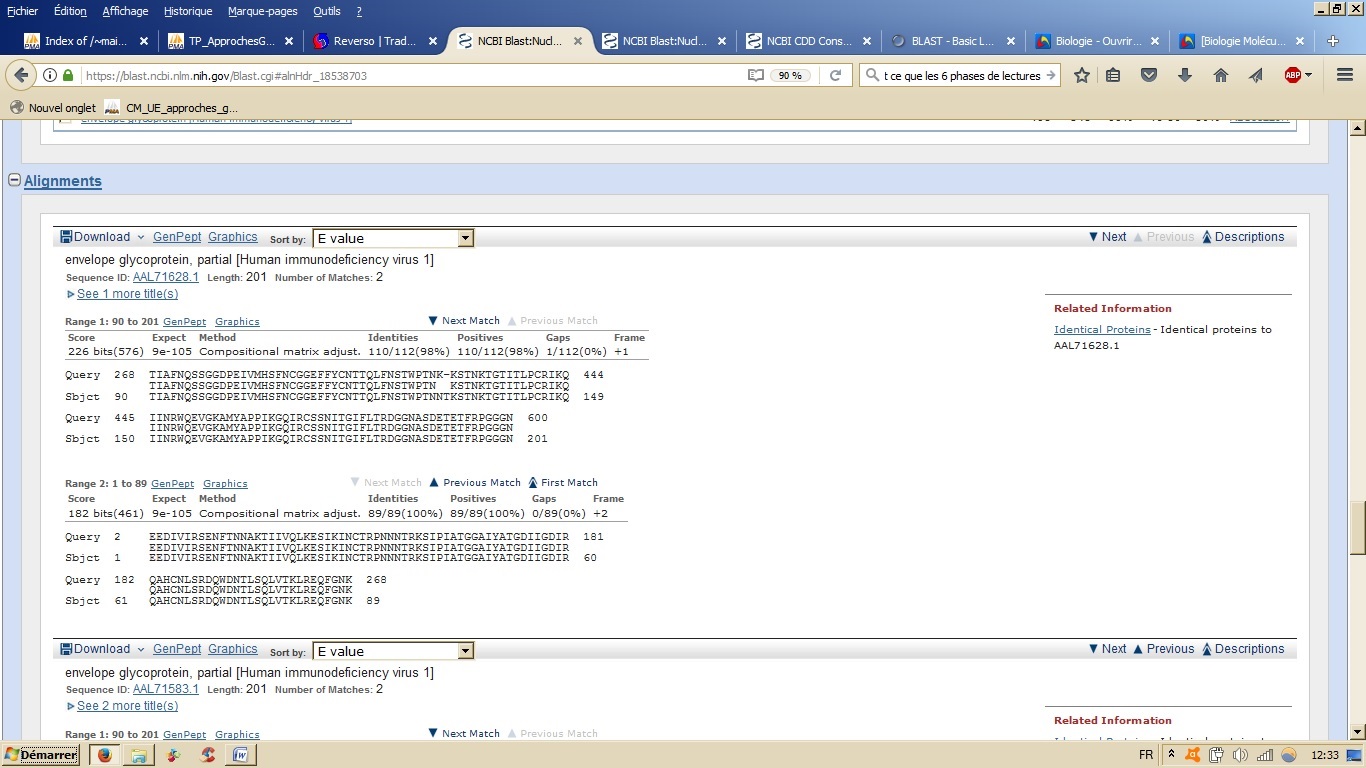
Comme premier exercice, j'ai copié une séquence d'ADN de 601 nucléotides dans BLASTx en utilisant la base de données nr. Il me trouve une similarité avec une e-value de 9e-105 pour une protéine de l'enveloppe glycoprotéique du HIV.
Le résultat de la requête donne ceci :
De BLAST x, je sais qu'il traduit notre séquence requête dans les 6 cadres de lecture avant de les comparer à la base de données.
1ère question : pour travailler sur les 6 cadres ouverts de lecture , BLAST recréé le brin complémentaire de la séquence requête?
2ème question : à quoi correspond le triplicata de la séquence protéique donnée en réponse?
Cordialement.
-----