Bonjour j'aurais une série de questions
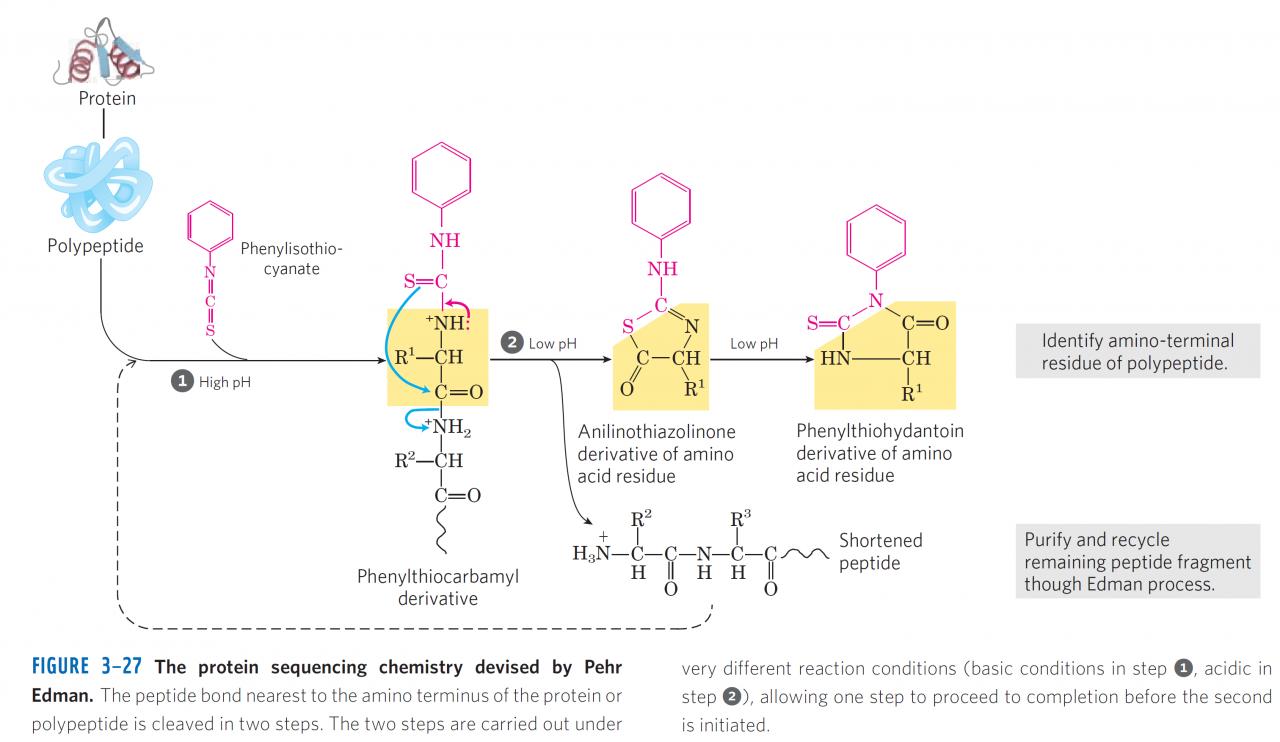
Supposons que l'on connait le nom d'une protéine dans un organisme et que l'on veut identifier la structure primaire pour déterminer quel gène la code.
Comment et avec quel protocole et matériel peut on concrètement faire ça ?
Je connais l’électrophorèse qui permet de différentier des protéines en fonction de leur masse et la spectrométrie de masse qui permet d'identifier les acides aminés présents.
Mais ma question principale est la suivante
Comment isoler une protéine voulu ? Identifier ensuite sa séquence en acide aminé ? Et enfin identifier la séquence de paire de base qui la code ?
Merci pour votre attention !
-----