Bonsoir,
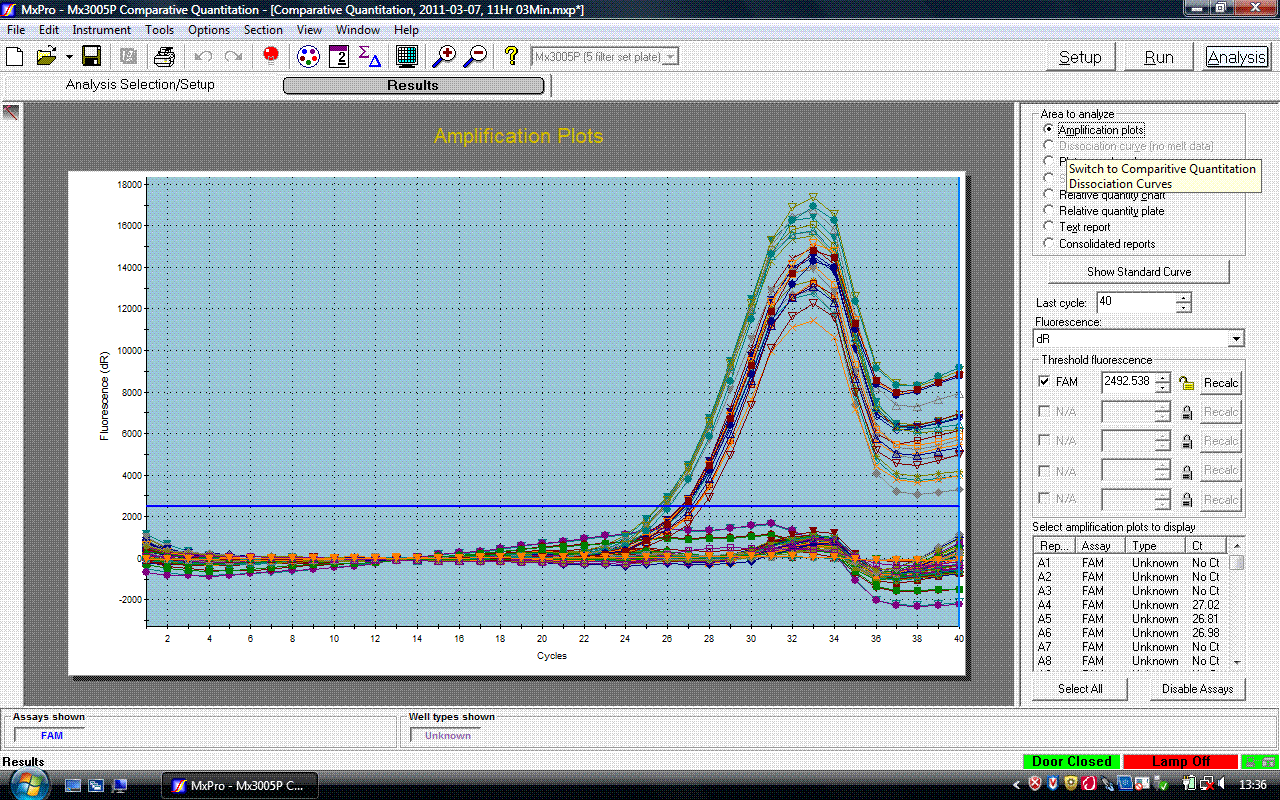
Je ss en stage de master et je fais des RTqPCR sur des micro ARN. J'ai un soucis avec mes courbes d'amplification, elles commencent bien lineaires mais a la fin au lieu d'avoir un plateau, les courbes font les montagnes russes. Je pensais que ça pouvais venir de la quantité d'ADNc que je met au depart qui est peut etre trop importante (40ng) et qui fini par saturer l'enzyme.
Si l'un de vous a l'habitude de ce genre d'experience et qu'il connait un peu le troubleshooting de ce type de manip je ss interessé. En effet je ne sais pas si je peux interpreter le Ct de ce type de courbes qui finissent bizarre.
Merci d'avance.
-----







 ?
? 
 .
. 

 Elle peut diminuer mais de manière prévisible et linéaire selon la température.
Elle peut diminuer mais de manière prévisible et linéaire selon la température.