Bonsoir,
Je ne suis pas certain de ma réponse pourriez vous me donner votre avis svp?Merci d'avance
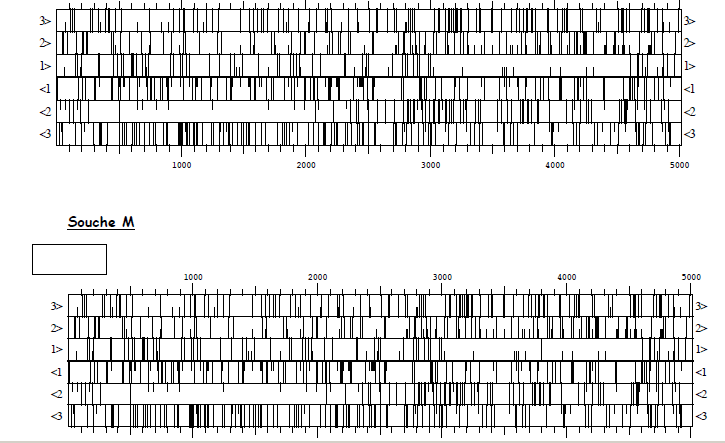
A partir d'une banque d'ADN génomique construite à partir de cellules de levure d’une
souche S de Schizosaccharomyces pombe, on a isolé un fragment d'ADN de 5 000
paires de bases (pdb). Ce fragment d'ADN est analysé à l’aide du logiciel DNA strider
sur micro-ordinateur Macintosh. La recherche de séquences pouvant correspondre à des
gènes codant des protéines a été faite en utilisant la fonction "ORF map" du logiciel qui
fait apparaître à l’écran de l’ordinateur une carte des « phases ouvertes de lecture »
(en anglais : Open Reading Frame, d’où l’abréviation ORF). On peut ainsi détecter les
CDS potentielles présentes sur ce fragment d’ADN.
Rappel : sur ce type de carte :
- un trait vertical court indique la présence d’un triplet 5’ATG3’
- un trait vertical long indique la présence d’un triplet 5’TAA3’, 5’TAG3’ ou 5’TGA3’
Quelle est la longueur en acides aminés des protéines synthétisées à partir
de ces CDS et leur masse moléculaire respective, en sachant que la masse
moléculaire moyenne d’un acide aminé est de 110 Dalton ?
Mon travai :
On a 5000pb=5000 nucléotides.Il faut 3 nucléotides pour former un acide aminé soit :
5000/3~1666 aa
(110 dalton/aa) x (1666 aa)=183260 dalton
1 nucléotide=0,34 nm
1aa = 3x0,34 =1,02 nm
d'où 183260 x 1,02 = 186925,2 nm = 0,186925,2 mm
Voila
-----