Bonjour à tous,
je suis actuellement en stage dans un laboratoire faisant des études sur le sol et j'ai un petit problème.
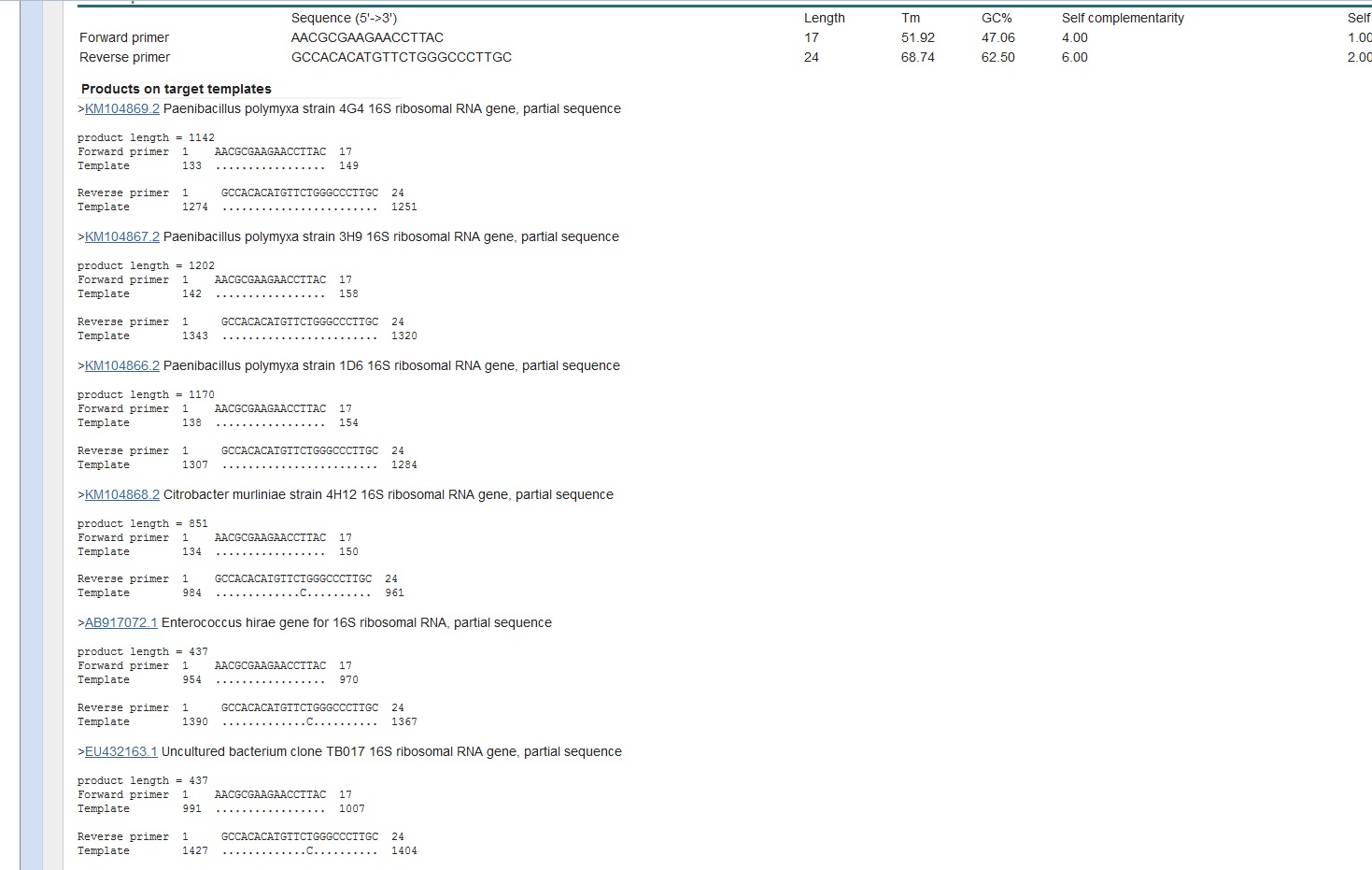
Je m'explique.. Nous avons réalisé une PCR sur nos échantillons de terre avec des amorces trouvé dans une revu scientifique correspondant en tout point à notre sujet, le problème étant qu'en attendant de pouvoir faire la DGGE ma maitre de stage m'a demandé de vérifier grace au site du NCBI et en faisant un blast que les séquences d'amorce matchaient bien avec les séquence ARNr 16s de bactérie présente généralement dans le sable et suceptible de sortir lors de la DGGE et d'en déduire leur taille. Cependant aucune ne matche correctement.
Le problème viens-t-il de ma mauvaise maitrise du blast ou bien du choix des amorces ?
amorce fwd : AACGCGAAGAACCTTAC
amorce reverse : GCCACACATGTTCTGGGCCCTTGC
et les bactéries que l'on trouve générallement dans le sol sont du genre : pseudomonas, rhizobium, azotobacter, nitrobacter, streptomyces
merci beaucoup d'avance si quelqu'un arrive à me sauver !!!!
-----