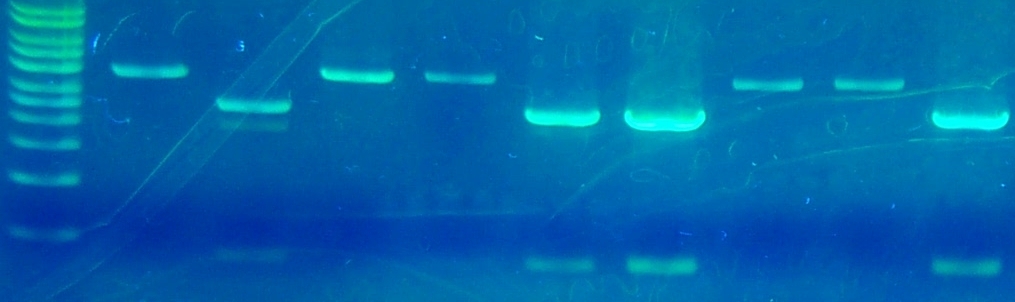
Bonjour,
je sollicite votre aide pour résoudre mon problème. Suite à un sous clonage, mon insert d'intérêt (environ 0.42kb) à été intégré dans un vecteur (3.6kb)... soit une taille totale après sous clonage de 4.02Kb. Après test de différentes colonies (9 en tout) et digestion de mes plasmides (double digestion enzymatique: XhoI / NcoI)...le résultat sur gel donne ceci:
La taille des poids moléculaires donnent de haut en bas: 10Kb, 8, 6, 4, 3, 2.5, 2, 1.5, 1 et 0.5Kb.
Observation et problème:
Mon fragment d"intérêt de 0.42Kb est présent pour les conditions n°2 - 5 - 6 et 9...mais la taille du vecteur ne corresponds pas (il fait entre 2 et 2.5Kb au lieu de 3.6Kb).
Or, les autres conditions ne possèdent pas mon insert, mais ont la taille du vecteur OK.
Comment cela se fait-il que les conditions contenant l'insert est un vecteur de taille inférieure à celui de départ?
Est ce que le sous-clonage est réussi ou non?
Merci d'avance pour vos éléments de réponse!
Cordialement.
-----