Bonjour à tous,
voilà je suis étudiant en M1 et j'effectue un stage en labo. J'essaie d'amplifier un gène (TBX3) de 2171 pb par PCR, la matrice est un plasmide de 8200 pb en comptant le gène (pT-Rex-DEST30). Je compte récupérer le produit de PCR pour clonage dans un autre vecteur.
Pour l'instant je bosse avec la Dream Taq DNA pol (mise au point de la PCR avant utilisation de la Phusion) en suivant le protocole du fabricant Thermo Scientific (mais en faisant mon mix, je n'ai pas le master mix de Thermo Sc.).
1) Voilà le mix pour un point :
- Dream Taq Buf. 10X 2,5 µL
- dNTP 0.5 µL (0.2 mM final pour chaque)
- DNA pol 0.125 µL (0.625 U)
- primer For/Rev 0.625 µL (250 nM chacun)
- matrice 2.5 µL (100 ng) (Thermo dit de ne pas dépasser 1 ng)
- eau qualité BM qsp. 25 µL
2)La PCR : 5 cycles à 60°C, 35 cycles à 65°C selon les recommandations du fabricant
3) Mes amorces :
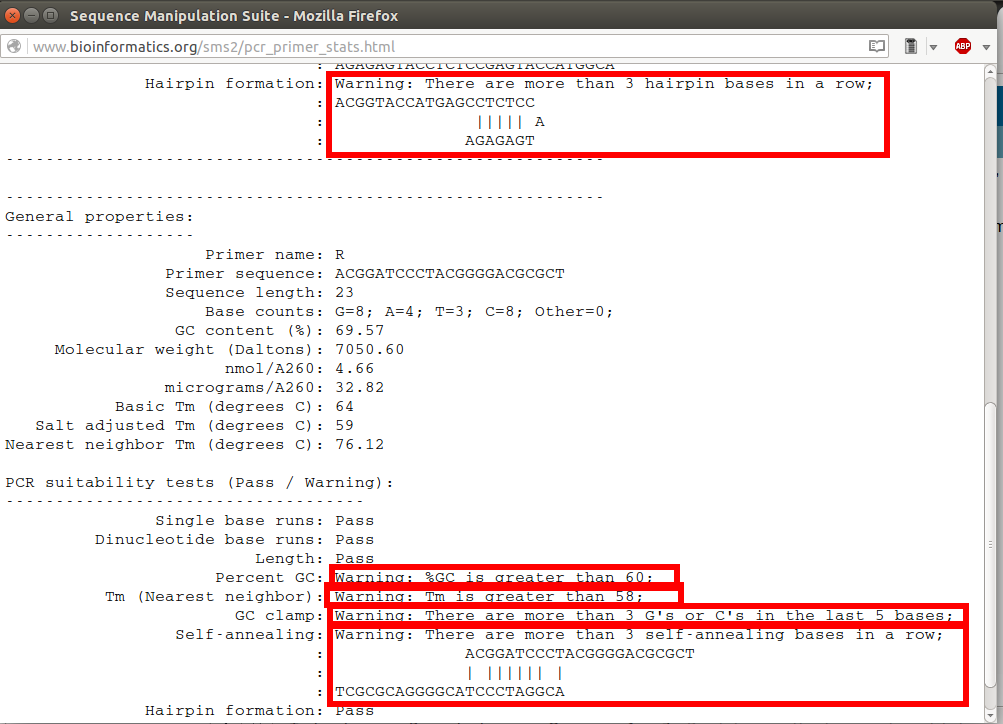
- For = ACGGTACC.ATG.AGC.CTC.TCC.ATG.AGA.GA (avec site KPN1) Tm = 60-65°C
- Rev = ACGGATCC.CTA.CGG.GGA.CGC.GCT (avec site BamH1) Tm = 65-70°C (selon les sources)
Mon problème (évident si il en est) ça marche pas !!!
J'ai fait varier:
- l' annealing de 50 à 70°C
- la concentration en MgCl2 (2 mM dans le Dream Taq buf. 1X à 3, 4,5 et 6 mM)
Après migration en gel agarose 1% j'ai pas mal de bandes aspécifiques (rien dans les contrôles).
Hier j'ai eu mon premier résultat à 3 mM de MgCl2, 60-65°C sur 40 cycles et 100 ng de matrice mais plus rien aujourd'hui.
A noter que l'augmentation de MgCl2 réduit pas mal l'intensité des bandes aspécifiques (bizarre nan ? c'est un peu l'inverse de la théorie)
Pour la petite histoire, je compte à terme (si la PCR marche haha !) cloner TBX3 dans un pcDNA3 puis le transfecter dans des cellules SiHa afin d'observer les changements éventuels sur l'expression des oncogènes E6/E7 produits par HPV16 (infectant les SiHa).
Merci d'avance pour vos précieux conseils !
-----




 ) essai d'amplifier ce tube (pas celui qui t'as servi a avoir ce résultat, mais bien le tube après PCR) et fais en deux tubes a PCR, un ou tu te servira directement 2.5µL de ce produit PCR dans tes 25µL et un ou tu diluera préalablement 50x ce produit et tu te servira 2.5µL du produit PCR dilué 50x (si ta bande était bien visible tu peux même diluer a 100)
) essai d'amplifier ce tube (pas celui qui t'as servi a avoir ce résultat, mais bien le tube après PCR) et fais en deux tubes a PCR, un ou tu te servira directement 2.5µL de ce produit PCR dans tes 25µL et un ou tu diluera préalablement 50x ce produit et tu te servira 2.5µL du produit PCR dilué 50x (si ta bande était bien visible tu peux même diluer a 100)